液滴生成技术使研究人员能够使用最小样本量进行大规模研究,这种方法可以使 DNA 测序更加高效
和经济。此外,它还可以帮助识别与癌症诊断相关的单细胞内微小变异。本文讨论了液滴生成在DNA 分析中的应用,并展示了使用液滴微流体进行实验所需的基本设置。
DNA 分析的机遇和挑战
科学家对 DNA 进行分析以识别癌症突变、表征人类抗体及区分微生物等。DNA 分析涉及多种技术,包括使用已知序列探针阵列或采用下一代测序(NGS)平台对基因组进行测序[1]。

NGS 平台利用芯片阵列实现大规模并行分析,可同时检测数亿个分子,从而降低成本并提高效率[1]。
然而,传统芯片阵列的分析能力受限,且需要额外的流体处理和试剂混合系统,增加了实验流程复杂度并限制了总吞吐量。为了降低每个碱基的测序成本,通常需要并行处理多个样本,而条形码标记虽然可以部分缓解此问题,却增加了实验流程的时间[1]。
基于液滴的微流体技术
微流控液滴技术是利用不可混溶的相(通常是水相和油相)生成离散液滴,并通过微流控泵高速精确
控制液滴的生成[3]。其主要优势包括:
- 低样本体积需求
- 高自动化程度
- 高通量
- 低成本

微流控设备能够快速、精准地控制流体体积,提高DNA 分析吞吐量。例如,液滴微流控技术可实现每秒成千上万个液滴的生成和分选[1]。每个液滴即为独立的反应室,适用于数字 PCR 分子计数、蛋白质结晶筛选和基因表达分析等应用。反应次数可随着液滴生成速率的增加而扩大,同时液滴体积的减小可降低试剂消耗,使实验更具灵活性[1]。
整体而言,该技术可在极少的试剂消耗下进行数亿次反应,极大提升实验效率并降低成本。然而与基于芯片阵列的固定点检测不同,液滴在微流控通道中流动,无法通过位置信息标记反应结果,因此需要额外的标记方法[1]。
高通量单细胞基因组测序
传统测序技术通常难以检测基因组中的微小变异,因此科学家利用单细胞全基因组测序来识别拷贝数变异和单核苷酸变异[2]。
通过微流控技术可实现高通量、可扩展的单细胞测序。例如,一项研究采用条形码标记技术,对单个癌细胞的扩增基因组DNA 进行分析。该方法能够在癌症完全缓解期检测到具有致病突变的细胞,并揭示急性髓系白血病(AML)肿瘤的复杂克隆进化过程,这些信息是传统测序方法无法捕获的[3]。因此,该技术有望提高癌症基因组异质性分析的精度,并优化个性化治疗策略[3]。
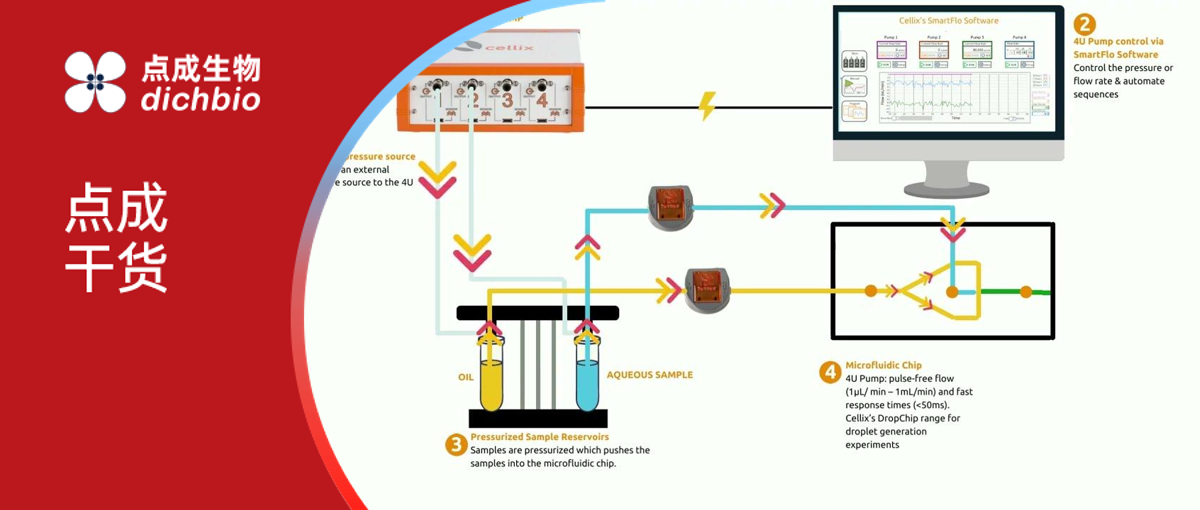
如何开始实验?
液滴微流控技术提供了一种高通量、低成本的 DNA 分析方式。如果您希望开展相关实验,以下是所需的基本设备:
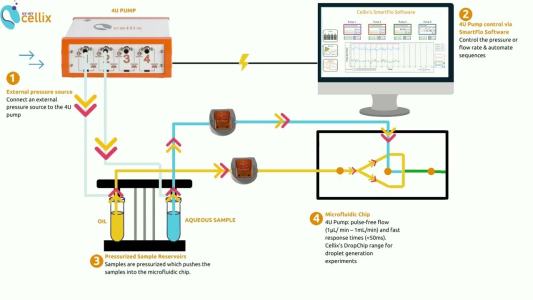
- 2-3 台微流控泵(或 1 台具备 2-3 个通道的泵,如 4U 泵):控制连续相(油相)和分散相(水相)的流动。推荐使用 4U 压力泵或 2-3 台 ExiGo 微流控注射泵,其中 4U 压力泵可独立控制 4个通道,实现稳定且精准的流量控制。
- 2-3 个流量传感器:实时监测油相和水相的流量,确保流体稳定。
- 微流控芯片:生成液滴并保证液滴尺寸的可控性。
- 稳定的通道表面化学修饰:提高液滴稳定性。
- 表面活性剂:稳定油水界面,增强液滴的稳定性。
- 油相:提高液滴稳定性。
- 管路:连接泵与微流控芯片。
点成 Cellix 可提供完整的液滴微流控实验套件或单独的组件。如需了解更多信息,请联系我们。
想要了解更多信息,请联系我们